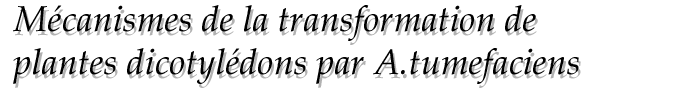
|
|
Insertion du plasmide T-DNA
Préparation
du plasmide T-DNA
Le plasmide T-DNA est initialement intégré dans le Ti-DNA. C'est sous forme de simple brin qu'il est exporté dans le noyau de la cellule végétale cible. Les protéines VirD1 et VirD2 sont des endonucléases spécifiques qui reconnaissent les bords du T-DNA (séquences spécifique de 25 paires de bases) ainsi que le brin qui sera exporté et l'excisent. VirD2 s'attachent de manière covalante sur l'extrémité 5' du simple brin du T-DNA. Le brin de T-DNA est recouvert par plusieurs copies (environ 600) de la protéine VirE2, ce qui permettrait la protection du T-DNA contre les nucléases de la cellule végétale cible (Howard et Citovsky, 1990). Les protéines virE1 stabilisent les protéines VirE2 (Citovsky et al., 1992, Sundberg et al., 1996). L'ensemble VirD2, VirE2 et T-DNA simple brin constitue le "compexe T". Des résultats récents suggèrent que les protéines VirE2 sont exportées séparément du T-DNA vers la cellule végétale (Lee1 et al., 1999}). On suppose que les protéines VirE2 et VirD2 sont aussi responsables de l'adressage du T-DNA vers le noyau, à travers les pores nucléaires. VirD2 aurait la structure d'une topoisomérase.
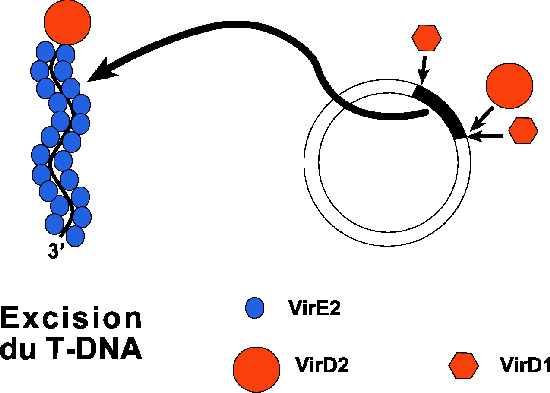
Transport du plasmide T-DNA Les protéines VirB (11 protéines produites par l'opéron virB) ainsi que VirD4 serviraient de canal entre la bactérie et la cellule végétale cible. VirB1 serait la seule des protéines VirB qui ne soit pas indispensable pour le transfert du T-DNA. De nombreuses études comparent le système de transfert du T-DNA d'Agrobacterium tumefaciens avec le système de conjugaison du plasmide F de E. coli, régulé par les gènes tra qui permettent l'établissement d'un tunnel entre la cellule hôte possédant le plasmide et une cellule cible dépourvue du plasmide. Ce "tunnel"est formé par un pilus par lequel transite le plasmide. Dans le cas d'Agrobacterium tumefaciens, la protéine VirB2 serait la composante essentielle du pilus qu'Agrobacterium tumefaciens forme pour se connecter sur la cellule végétale cible. La structure prédite de VirB2 est la même que TraA, impliquée dans la conjugaison du plasmide F de E. coli. virB2, virB3 et virB9 sont impliquées dans la synthèse du pilus de conjugaison (Jones et al., 1996). L'export de VirB2 nécessiterait l'action d'autres protéines Vir (Lai et Kado, 1998). VirB1, située sur la membrane interne d'Agrobacterium tumefaciens et ayant une structure N-terminale similaire aux transglycosidases bactériennes, serait responsable de la lyse local de la paroi cellulaire de la bactérie, permettant la formation du pilus (Baron et al., 1997). VirB1 est aussi responsable de la synthèse d'un propilin, impliqué dans le contact initial avec la cellule cible, par l'assemblage de protéines VirB1*, sécrété dans le milieu de culture (Baron et al., 1997, Chumakov et Kurbanova, 1998). La protéine chromosomale AcvB jourait un rôle essentiel dans le transfert du plasmide, mais ce rôle pourrait être remplacé par virJ, régulé par l'acétosyringone (Pan et al., 1995). VirB8 est située sur la membrane interne d'Agrobacterium tumefaciens (Thorstenson et Zambryski, 1994).
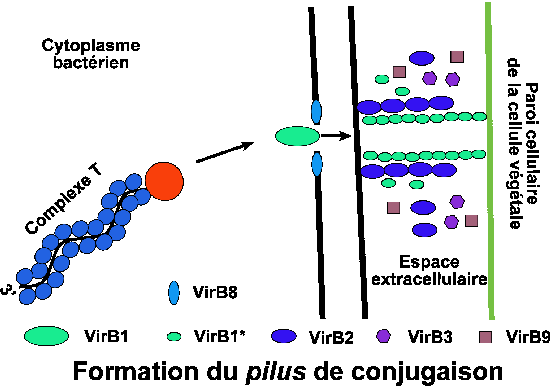
Intégration dans le génome végétal nucléaire L'intégration du plasmide au sein du génome de la cellule cible ne nécessite aucune protéine codée par le T-DNA. Elle ne dépend que des protéines bactériennes ainsi que des protéines de la cellule végétale chargée de réparer l'ADN. Plusieurs copies du T-DNA peuvent se retrouver dans le génome nucléaire de la cellule végétale infectée. L'intégration du T-DNA au sein du génome nucléaire se fait au hasard : aucune préférence pour un chromosome particulier n'a été observé dans les espèces couramment utilisées en recherche en biologie végétale. Il semble de la même manière qu'aucune structure génique ne soit favorisée lors de l'intégration du T-DNA. L'insertion du T-DNA dans l'ADN végétal nucléaire s'accompagne de petites délétions (13-73 paires de bases) au niveau du site d'insertion. D'autre part, on observe la copie d'une séquence de 158 paires de base situé à proximité du site d'insertion chez Nicotiana tabacum (Gheysen et al., 1987). Le côté 3' du T-DNA est très peu conserve après insertion alors que le côté 5' qui est lié à la protéine VirD2 est parfaitement conservé. Ces modifications suggèrent que l'intégration du T-DNA se fait en plusieurs étapes et que des enzymes impliquées dans la réparation de l'ADN végétal telles que ADN-polymérases, ligases, topoisomérases et hélicases. Ceci est fortement appuyé par le fait que deux lignés mutantes d'Arabidopsis hypersensibles aux radiations dans les ultraviolets et rayons gamma ne sont pas capable d'intégrer le T-DNA.
|
|
© 2000 Cyril Tikhomiroff $Id: partie3.html,v 1.2 2002/10/27 07:29:55 tikho Exp $ | |